Retours vers :
[Accueil]
[Sommaire du dossier]
JF Perrin mise à jour 2004-2014
[A propos de l'auteur]
[Droits de copie]
Dans le monde des bactéries (Eubacteria), on rencontre deux grands types d'enveloppes qui correspondent à deux grandes branches phylogéniques : le bactéries monodermes (dites aussi à paroi "gram-positive") et les bactéries didermes à LPS (dites aussi à paroi "gram-negative"). Les monodermes seraient plus "ancestraux".
Chez ces bactéries, la membrane cytoplasmique est entourée d'une enveloppe essentiellement constituée par une trame multicouches d'un peptidoglycane appelé muréine. C'est la paroi qui va donner à la bactérie sa forme et sa résistance à la pression osmotique. La paroi de peptidoglycane participe à régler la diffusion des nutriments vers la membrane cellulaire. Les figures suivantes permettent de comprendre la structure d'une paroi de bactérie "gram-positive" classique.
|
|
Et ci dessous, une figure plus chimique montrant la structure exacte de la N-acetyl-glucosamine (NAG) et de l'acide N-acetyl-muramique (NAM) et leur liaison en β-1-4 :
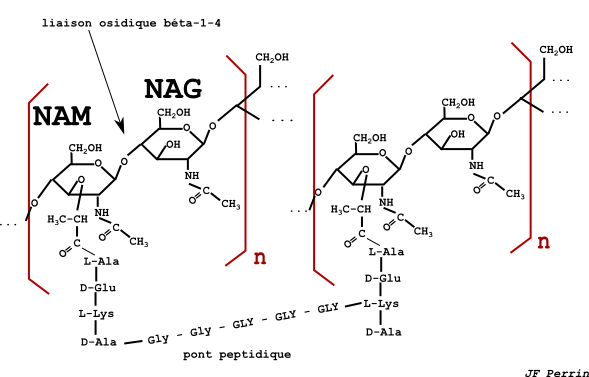
On retiendra les chaînes NAM-NAG et ses liaisons β1-4, les pontages peptidiques et la présence d'acides aminés de la série D dans les ponts peptidiques !
En comprenant que la muréine forme une enveloppe multicouches, on peut se faire l'image suivante de la paroi monoderme :
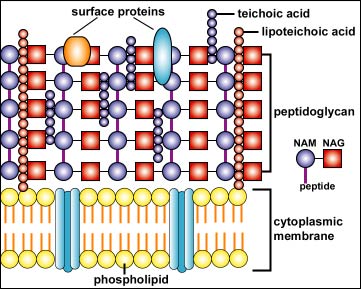 |
The Gram-positive cell wall appears as dense layer typically composed of numerous rows of peptidoglycan, and molecules of lipoteichoic acid, wall teichoic acid and surface proteins. Cette figure à gauche est issue de http://faculty.ccbcmd.edu/courses/bio141/lecguide/unit4/innate/u1fig9b.html. |
Il reste donc à savoir ce que sont les acides lipoteichoïques et téchoïques. Ce sont des glycopolymères intégrés dans les couches de muréine et ceci est bien expliqué par Christopher Weidenmaier et Andreas Peschel dans Nature Reviews Microbiology 6, 276-287 (April 2008):
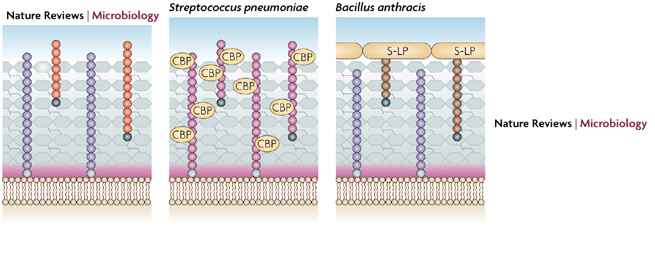
CWG polymers (cell-wall-glycopolymers) are shown as chains of circles within the cell wall. Differences in the composition of CWG repeating units are indicated by different colours. Linkage units that connect CWGs with peptidoglycan or lipids are shown as dark- or light-grey circles, respectively. See Fig. right for compositional details. CWGs connect choline-binding proteins (CBPs) in S. pneumoniae, S-layer proteins (S-LPs) in B. anthracis and mycolic acids in M. tuberculosis. Bacilli and mycobacteria often contain more than the two types of CWG shown here. http://www.nature.com/nrmicro/journal/v6/n4/fig_tab/nrmicro1861_F1.html |
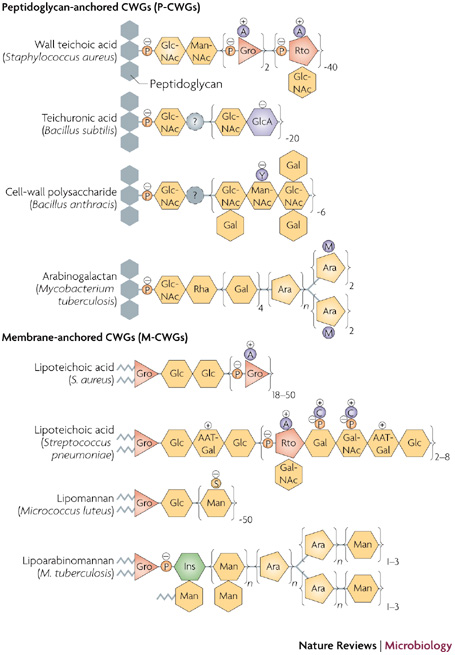
Trioses, peptoses and hexoses are shown as triangles, pentagons and hexagons, respectively. Sugars, sugar-derived alcohols and sugar-derived acids are shown in yellow, orange and purple, respectively. Fatty acids are shown as zigzag lines. Many structural details, such as sugar-bonding patterns, are not included. N-acetylglucosamine (GlcNAc) at the first position of B. subtilis teichuronic acid is thought to be most likely, as the tagO gene, which is responsible for the incorporation of GlcNAc at this position, has been shown to be indispensable for teichuronic acid biosynthesis. A GlcNAc residue at position one of B. anthracis P-CWG is also thought probable because of the presence of tagO in the B. anthracis genome, although no experimental evidence is available to confirm this. Unclear second positions in P-CWG linkage units are indicated by question marks. The exact position of pyruvylation in B. anthracis P-CWG is unknown. Mycobacterial CWGs are more extensively branched than indicated in this simple schematic. Non-glycosyl residues: A, D-alanine; C, choline; M, mycolic acid; P, phosphate; S, succinate; Y, pyruvate. Glycosyl residues: AAT-Gal, 2-acetamido-4-amino-2,4,6-trideoxy-D-galactose; Ara, arabinose; Gal, galactose; GalNAc, N-acetylgalactosamine; Glc, glucose; GlcA, glucuronic acid; Gro, glycerol; Ins, inositol; Man, mannose; ManNAc, N-acetylmannosamine; Rha, rhamnose; Rto, ribitol. http://www.nature.com/nrmicro/journal/v6/n4/fig_tab/nrmicro1861_F2.html |
Le peptidoglycane est très très généralement multicouches et c'est lui qui conduit ainsi à l'effet "Gram+" à la coloration de Gram : la décoloration à l'alcool (ou au mélange acétona-alcool) après la première étape de coloration au cristal violet puis de mordançage à l'iode ioduré est plus lente que dans la cas des bactéries didermes ou il n'y a que très peu de couches de peptidoglycane.
Mais cette propriété n'est pas générale. Certaines bactéries monodermes à paroi pas très épaisse se laissent facilement décolorer et apparaîtront roses (Gram-) après la contre-coloration à la safranine lors d'une coloration classique de Gram. C'est fréquent chez le genre Bacillus. Les Mycobactéries (voir ci-dessous) sont des monodermes qui prennent très difficilement les colorants dans la coloration de Gram ! Les Mollicutes sont des bactéries sans paroi, en fait des monodermes ayant perdu leur paroi, colorées "au Gram", elles seraient Gram- !
La figure "Enveloppe des corynebacterineae" ci-dessous montre clairement le cas de ce sous-ordre de bactéries : une double couche lipidique à acides mycoliques vient se rajouter à la face externe des couches de muréine qui sont bourrées d'acides lipoteichoïques. On a donc là l'invention d'une 2° membrane externe mais sans lipides de type LPS comme on le trouve chez les didermes classiques. Cette membrane externe à mycolates conduit à une paroi très hydrophobe et conférant une forte résistance aux macrophages chez les espèces pathogènes (propriété majeure si on pense à Mycobacterium tuberculosis et à la tuberculose) ...
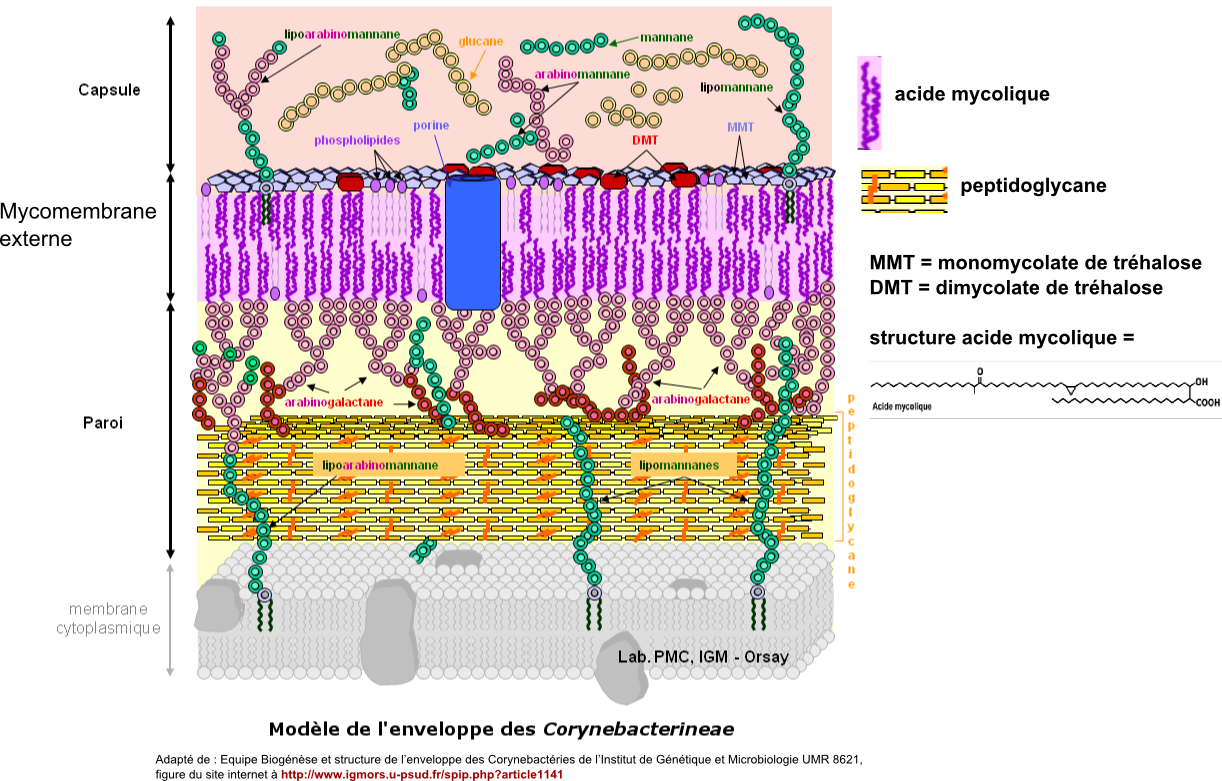
Au laboratoire, les mycobactéries ne vont quasiment pas prendre la coloration de Gram, même après fixation, leur paroi demeure trop "étanche". Pour les colorer, on "force" une coloration à la fushine (à chaud, ou très très longtemps) puis on décolore avec un mélange acide-alcool qui va décolorer toutes les bactéries sauf les mycobactéries qui vont retenir leur coloration rose à la fushine grâce à leur paroi super "étanche". On finit par une petite coloration au bleu de méthylène : les mycobactéries seront roses (colorées à la fushine et pas décolorées), les autres bactéries apparaissent bleues (colorées à la fushine puis décolorées puis colorées en bleu).
Pour les esthètes, voici la structure de base d'un acide mycolique : ![]()
Les bactéries de la grande famille des mollicutes (à "peau molle") sont des bactéries dépourvues de paroi. Il s'agit en fait de monodermes ayant perdu leur paroi. Ces bactéries sont des parasites d'organismes eucaryotes. Elles sont bien connues de tous ceux qui travaillent sur des cultures eucaryotes et qui cherchent à éviter les contaminations par les toutes petites bactéries parasitaires sans paroi de type mycoplames (elles passent à travers les filtres de porosité 0,2 µm!).
Le lysozyme qui hydrolyse les liaisons β-1-4 entre la N-acetyl-glucosamine (NAG) et l'acide N-acetyl-muramique (NAM) de la muréine est très utilisé au laboratoire pour "digérer" les parois bactériennes.
La paroi peptidoglycane est en perpétuel remaniement. Une dynamique indispensable pour les phénomènes de division et de croissance en taille et qui détermine la forme des bactéries.
La division bactérienne est traitée à http://www.perrin33.com/microbiologie/lereste/divisiondesbacteries.html
La synthèse du peptidoglycane de paroi et son remaniement dynamique constant fait appel à des peptidases (carboxypeptidases) et des transpeptidases. Celles-ci sont les cibles des antibiotiques de la famille des β-lactames (dont les célèbres pénicillines). La biosynthèse du peptidoglycane et les antibiotiques β-lactames son traités à http://www.perrin33.com/microbiologie/lereste/lactam_2.php.